相关疑难解决方法(0)
PyTables与h5py之间的HDF文件格式兼容性
几周前,我开始在Python上使用HDF文件格式,当您这样做时,您首先意识到的是,有两个主要的库,尽管它们略有不同,但它们都很棒:Pytables(与用于可视化的ViTables工具配合使用)和h5py(与HDFView和HDFCompass一起很好地工作)。
可悲的是,这两个库不能很好地配合。我在几个地方(如here或here)都读到,其想法是通过以这种方式将它们一个“放置”在另一个上来使它们兼容:
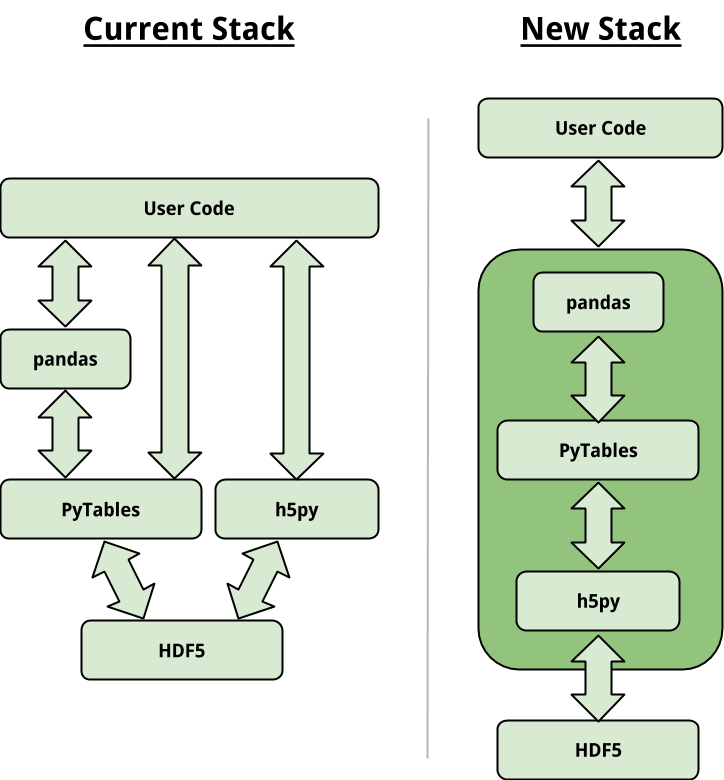
这是在SciPy 2015上讨论的,所以我的问题是:有人知道这是怎么回事吗?目前情况如何?
6
推荐指数
推荐指数
1
解决办法
解决办法
924
查看次数
查看次数
读取行的最佳HDF5数据集块形状
我有一个合理的大小(压缩后的18GB)HDF5数据集,并希望优化读取行的速度。形状为(639038,10000)。我将多次读取整个数据集中的选定行(例如〜1000行)。所以我不能使用x:(x + 1000)来切片行。
使用h5py从内存不足的HDF5中读取行已经很慢,因为我必须传递一个排序列表并求助于高级索引。有没有一种方法可以避免花式索引,或者我可以使用更好的块形状/大小?
我已经阅读了一些经验法则,例如1MB-10MB的块大小,并且选择了与我所读内容一致的形状。但是,构建大量具有不同块形状的HDF5文件进行测试在计算上非常昂贵且非常缓慢。
对于每个〜1,000行的选择,我立即将它们求和以获得长度10,000的数组。我当前的数据集如下所示:
'10000': {'chunks': (64, 1000),
'compression': 'lzf',
'compression_opts': None,
'dtype': dtype('float32'),
'fillvalue': 0.0,
'maxshape': (None, 10000),
'shape': (639038, 10000),
'shuffle': False,
'size': 2095412704}
我已经尝试过的东西:
- 用块形状(128,10000)重写数据集(据我估计约为5MB)太慢了。
- 我看了dask.array进行了优化,但是由于〜1,000行很容易容纳在内存中,所以我看不到任何好处。
2
推荐指数
推荐指数
1
解决办法
解决办法
3105
查看次数
查看次数